Segmentation des compartiments cellulaires
Segmentation adaptée à chaque organite
Chaque compartiment cellulaire a une morphologie propre, une structure fibreuse ne peut donc pas être segmentée de la même manière qu’un spot (kinétocore) ou une structure ronde (noyau). La détection d’un compartiment est un processus complexe qui nécessite de paramétrer finement les outils de segmentation pour récupérer correctement le signal fluorescent.
D’une manière générale, ces groupes de composants se segmentent de la même manière (liste non exhaustive) :
- Fibres : filaments d’actine, microtubules, filaments intermédiaires, matrice extracellulaire
- Compartiments complexes : appareil de Golgi, réticulum endoplasmique, chromosomes durant la mitose, fuseau mitotique, mitocondries, ribosome
- Spots : endosomes, lysosomes, MOC, vacuoles, vésicules
- Membrane : membrane cytoplasmique, membrane nucléaire
Exemples de segmentations adaptées du centrosome, du Golgi et du noyau
La segmentation des compartiments permet d’analyser leur morphologie (taille, forme, intensité, position dans la cellule) et leur position par rapport aux autres compartiments.
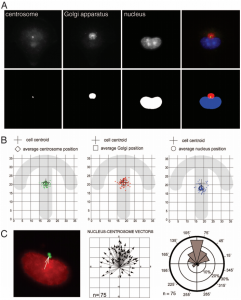
La figure suivante illustre la position des compartiments dans des cellules adhérentes sur des micropatterns. Les trois compartiments cellulaires sont segmentés séparément : le centrosome, l’appareil de Golgi et le noyau. La figure A montre chaque compartiment et sa segmentation. La figure B montre les distributions des compartiments dans l’espace (centrosome en vert, Golgi en rouge et noyau en bleu). La figure C représente les vecteurs noyaux-centrosomes et leurs directions.
Références
Anisotropy of cell adhesive microenvironment governs cell internal organization and orientation of polarity.Théry M, Racine V, Piel M, Pépin A, Dimitrov A, Chen Y, Sibarita JB, Bornens M. Proc Natl Acad Sci U S A, 103(52):19771-6, 2006.
Intéressé par des logiciels ou services ?
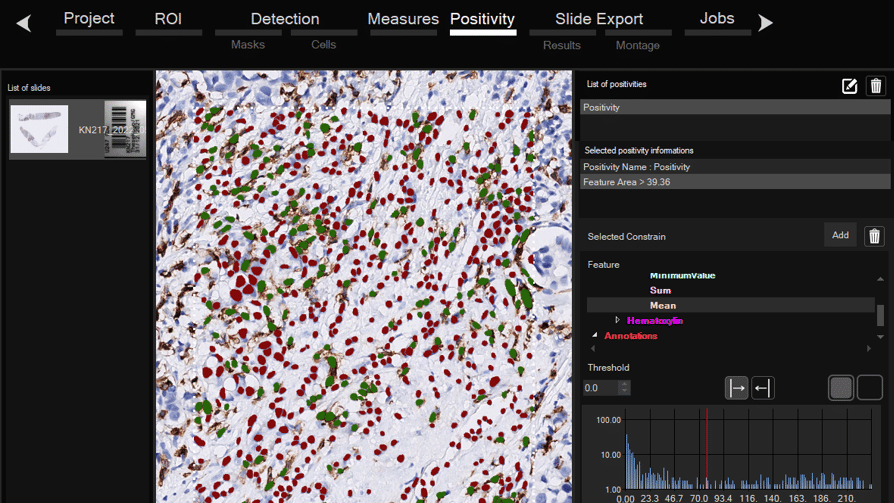
Logiciel pour échantillons histologiques
Détecte et quantifie automatiquement et facilement vos lames histologiques, sans nécessiter d’expertise en bio-informatique.
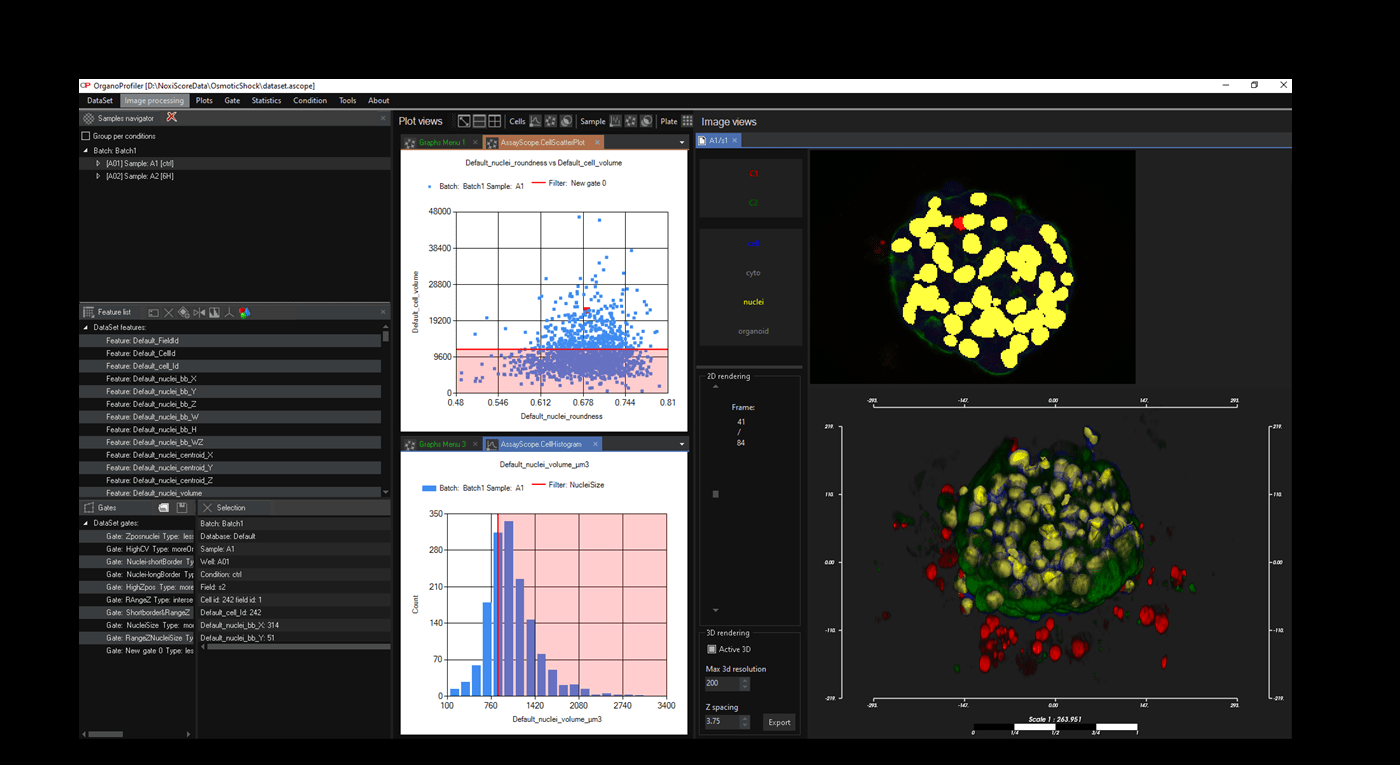
Logiciel pour organoïdes, sphéroïdes, cultures cellulaires et tissus 3D
Permet la segmentation 3D et l'analyse de criblage à haut contenu pour naviguer dans vos essais et étudier l'effet des médicaments
Service d'analyse et de caractérisation
- Analyse d'image d'études cliniques
- Caractérisation des effets de médicaments
- Détection automatique d'événements rares

Développement de logiciel pour l'imagerie biomédicale
- Solutions customisées avec une ergonomie UIX
- IA pour automatisation
- Industrialisation des analyses en laboratoires
QuantaCell, Hôpital Saint Eloi, IRMB
Contact
+33 (0) 9 83 33 81 90
80 av Augustin Fliche
34090 Montpellier, France