Croisement d’informations hétérogènes
Fusions de données de microarray avec des informations publiques de bio-informatique
La masse d’information accessible par internet sur les données biologiques et chimiques est énorme. Ces informations peuvent être utilisées pour enrichir les résultats expérimentaux.
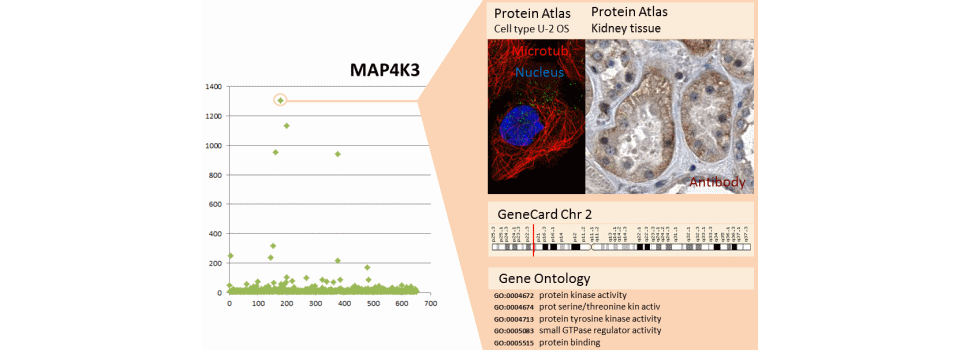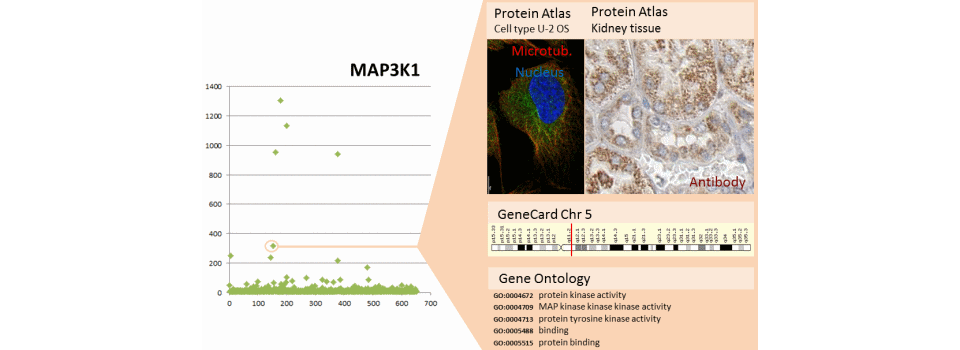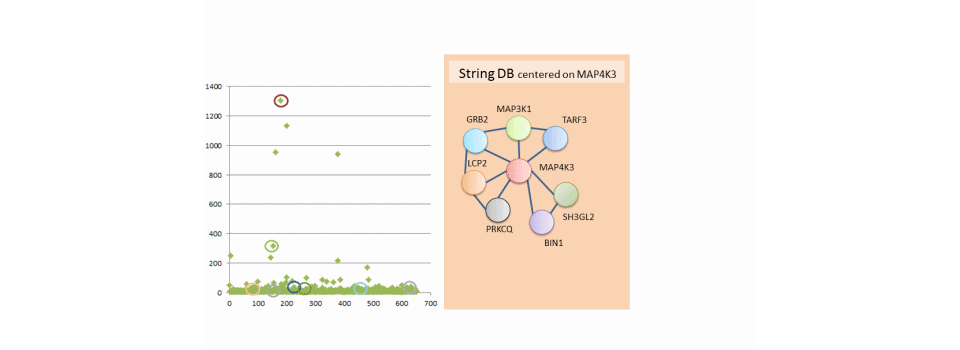
L’exemple présente les résultats d’un microarray du kinome complétés par des données publiques. Les données du microarray sont présentées à gauche. L’utilisateur sélectionne un gène (MAP4K3 puis MAP3K1) et les données publiques apparaissent dans les fenêtres à droite afin de croiser les information utilisateur avec les informations publiques.
Accès aux informations biomédicales dans les bases de données publiques
Des informations publiques des gènes sélectionnées sont récupérées automatiquement à partir de plusieurs bases de connaissances publiques :
- GeneCard (http://www.genecards.org/)
- Gene Ontology (accédé par GeneCard)
- HumanProtein Atlas (http://www.proteinatlas.org/) donne la localisation des protéines dans les modèles cellulaires, dans les tissus de nombreux organes cancéreux ou sains
- EMAGE (http://www.emouseatlas.org/) fournit les expressions des protéines dans les embryons de souris
- STRING (http://string-db.org/) donne les données des intéractomes
De la même manière, l’intéractome de MAP4K3 est affiché dans la fenêtre de droite et chaque gène de l’intéractome sont sélectionnées dans les données du microarray.
Ces informations sont intégrées dans les interfaces graphiques des logiciels pour permettre une interactivité optimale.
Cette stratégie permet à l’utilisateur d’avoir un maximum d’éléments et de mieux interpréter le résultat de l’expérience.
Intéressé par des logiciels ou services ?
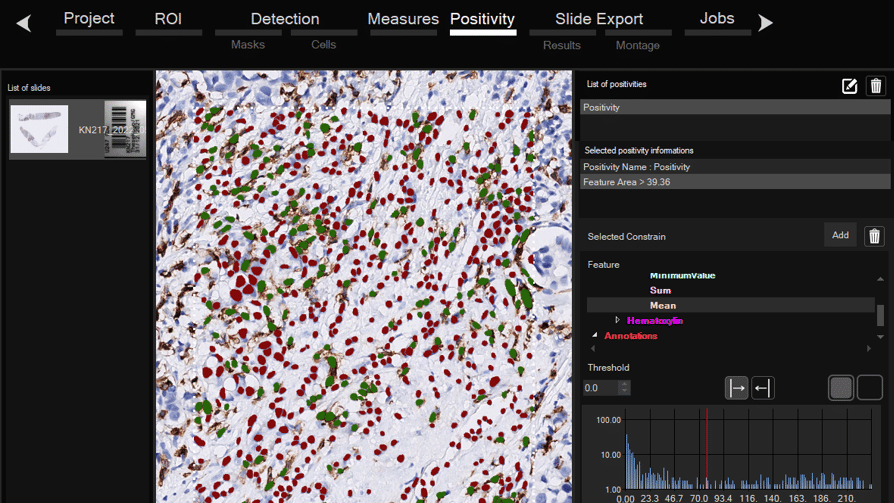
Logiciel pour échantillons histologiques
Détecte et quantifie automatiquement et facilement vos lames histologiques, sans nécessiter d’expertise en bio-informatique.
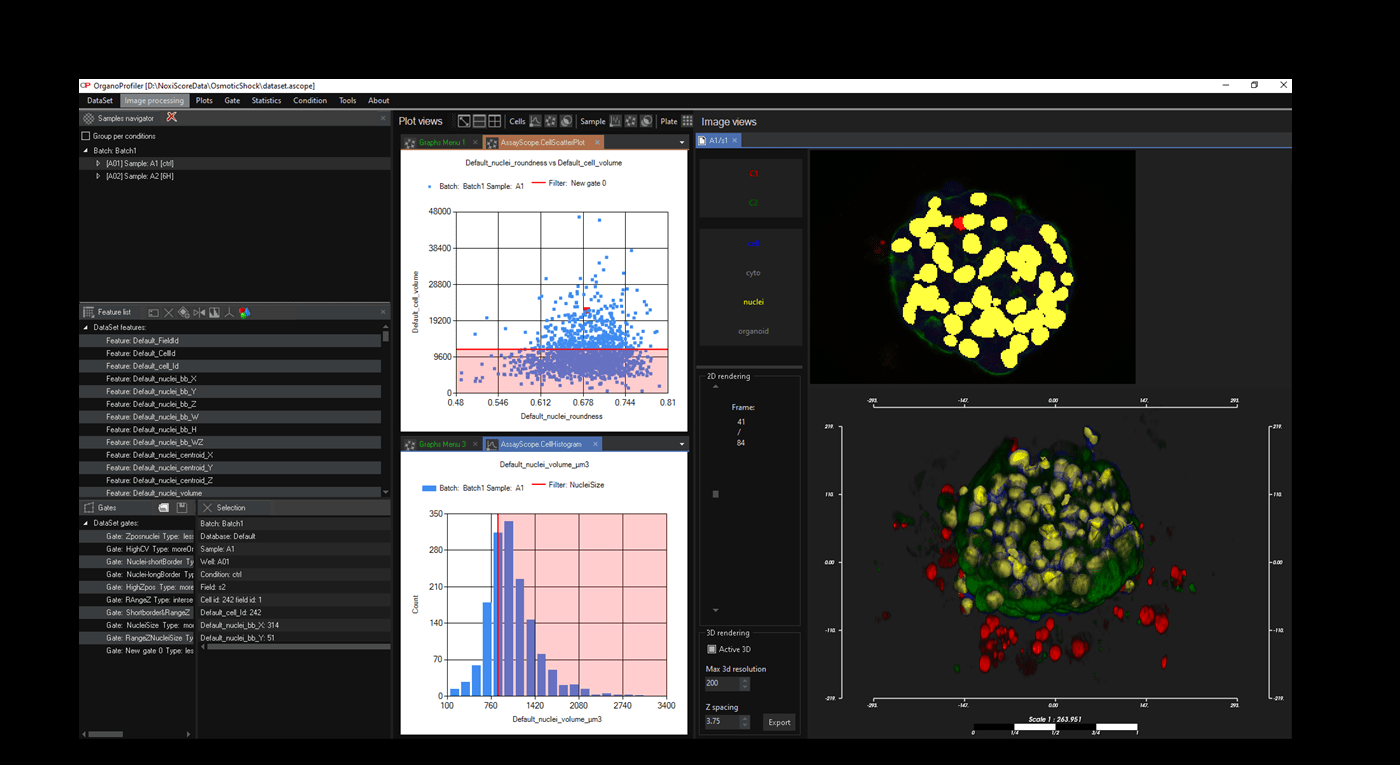
Logiciel pour organoïdes, sphéroïdes, cultures cellulaires et tissus 3D
Permet la segmentation 3D et l'analyse de criblage à haut contenu pour naviguer dans vos essais et étudier l'effet des médicaments
Service d'analyse et de caractérisation
- Analyse d'image d'études cliniques
- Caractérisation des effets de médicaments
- Détection automatique d'événements rares

Développement de logiciel pour l'imagerie biomédicale
- Solutions customisées avec une ergonomie UIX
- IA pour automatisation
- Industrialisation des analyses en laboratoires
QuantaCell, Hôpital Saint Eloi, IRMB
Contact
+33 (0) 9 83 33 81 90
80 av Augustin Fliche
34090 Montpellier, France